本案例共有羊驼免疫、噬菌体文库构建、噬菌体表面展示筛选,以及Sanger测序获得阳性VHH序列四个步骤。
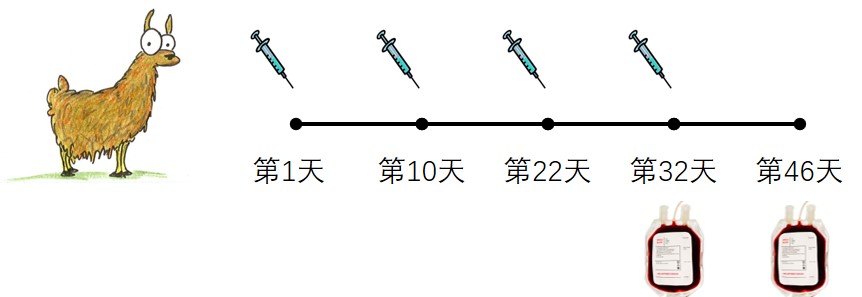
一、羊驼免疫:使用纯化的蛋白IL-18对Alpaca进行动物免疫
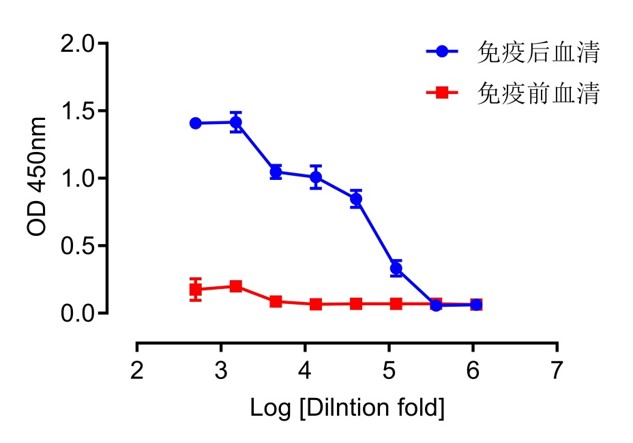
| 稀释比例 | 免疫后血清 | 免疫前血清 | ||||
| 500 | 1.429 | 1.43 | 1.367 | 0.263 | 0.156 | 0.105 |
| 1500 | 1.333 | 1.474 | 1.438 | 0.169 | 0.204 | 0.224 |
| 4500 | 1.046 | 1.097 | 1.001 | 0.095 | 0.076 | 0.087 |
| 13500 | 0.963 | 0.956 | 1.105 | 0.068 | 0.068 | 0.061 |
| 40500 | 0.893 | 0.775 | 0.874 | 0.067 | 0.067 | 0.072 |
| 121500 | 0.361 | 0.265 | 0.369 | 0.066 | 0.068 | 0.073 |
| 364500 | 0.061 | 0.05 | 0.06 | 0.075 | 0.075 | 0.06 |
| 1093500 | 0.059 | 0.064 | 0.065 | 0.054 | 0.069 | 0.070 |
二、噬菌体文库构建
电转化后文库总量:约7.30×108 cfu 。电转化后、扩增前留取的微量样本,扩增培养后从中随机挑取48个单克隆,进行下述实验:
PCR评估噬菌体载体片段插入率 使用特异性引物PCR扩增48个单克隆的VHH基因片段,琼脂糖凝胶电泳发现其中4个单克隆未正确插入VHH基因片段。
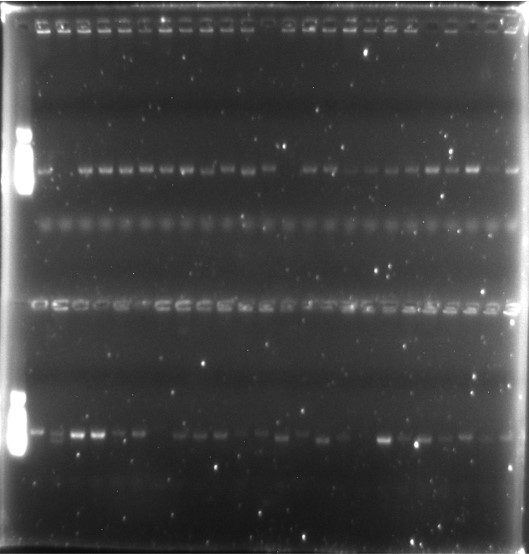
片段插入率:44/48 × 100% = 91.67%
Sanger测序评估序列多样性
获得DNA序列后,翻译为氨基酸序列,剔除测序失败、无效、重复的序列,使用MAFFT进行多序列比对后,最终获得38条CDR3区域完全不同的VHH片段(见下图)。

文库CDR3多样性:7.30×108 ×38/48 = 5.78×108 cfu
三、噬菌体表面展示筛选
第一轮初筛:共挑取了4块96孔板的微生物单克隆进行ELISA筛选,详细实验数据见下表格。
结论:第一轮初筛获得5个潜在阳性单克隆
plate 1
| / | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | 0.0707 | 0.0735 | 0.0791 | 0.0756 | 0.0751 | 0.0728 | 0.0735 | 0.074 | 0.0714 | 0.0746 | 0.0717 | 0.0799 |
| B | 0.1635 | 0.0763 | 0.075 | 0.0823 | 0.0808 | 0.0803 | 0.0746 | 0.0751 | 0.0773 | 0.0769 | 0.0706 | 0.0707 |
| C | 0.0741 | 0.0748 | 0.0747 | 0.0758 | 0.0887 | 0.0835 | 0.0766 | 0.0759 | 0.0753 | 0.0778 | 0.0735 | 0.0717 |
| D | 0.0714 | 0.0777 | 0.0791 | 0.0749 | 0.0783 | 0.0782 | 0.0773 | 0.0773 | 0.0776 | 0.0806 | 0.0759 | 0.0716 |
| E | 0.0738 | 0.0752 | 0.074 | 0.077 | 0.0743 | 0.0748 | 0.0762 | 0.0786 | 0.0758 | 0.0756 | 0.0788 | 0.0746 |
| F | 0.0795 | 0.0811 | 0.0819 | 0.0738 | 0.0827 | 0.0773 | 0.0771 | 0.0769 | 0.0731 | 0.0808 | 0.074 | 0.074 |
| G | 0.0734 | 0.0729 | 0.0732 | 0.0756 | 0.077 | 0.0743 | 0.0801 | 0.0799 | 0.0805 | 0.0808 | 0.0731 | 0.0642 |
| H | 0.0692 | 0.0729 | 0.0732 | 0.0741 | 0.0785 | 0.0793 | 0.0795 | 0.0778 | 0.0739 | 0.0731 | 0.0735 | 0.1851 |
plate 2
| / | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | 0.0712 | 0.0788 | 0.0778 | 0.0757 | 0.0766 | 0.0762 | 0.0822 | 0.0822 | 0.0764 | 0.077 | 0.0739 | 0.0883 |
| B | 0.0784 | 0.0754 | 0.0861 | 0.0732 | 0.0767 | 0.0921 | 0.077 | 0.0837 | 0.0773 | 0.0735 | 0.074 | 0.0786 |
| C | 0.0864 | 0.0818 | 0.0775 | 0.078 | 0.078 | 0.079 | 0.0781 | 0.0771 | 0.0825 | 0.0782 | 0.0804 | 0.0782 |
| D | 0.0784 | 0.1443 | 0.0788 | 0.0837 | 0.0801 | 0.0771 | 0.075 | 0.0814 | 0.0818 | 0.0971 | 0.0818 | 0.0761 |
| E | 0.0726 | 0.0778 | 0.0784 | 0.0843 | 0.0889 | 0.078 | 0.0781 | 0.0779 | 0.0772 | 0.0758 | 0.0757 | 0.0777 |
| F | 0.0769 | 0.079 | 0.0789 | 0.0827 | 0.0811 | 0.0783 | 0.0803 | 0.0836 | 0.0883 | 0.0846 | 0.0828 | 0.0817 |
| G | 0.0722 | 0.0792 | 0.0788 | 0.0767 | 0.0819 | 0.0839 | 0.0777 | 0.0766 | 0.0778 | 0.083 | 0.0753 | 0.0704 |
| H | 0.0735 | 0.0768 | 0.0798 | 0.0805 | 0.0756 | 0.0758 | 0.0738 | 0.0768 | 0.0807 | 0.0832 | 0.0812 | 0.189 |
plate 3
| / | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | 0.0694 | 0.0757 | 0.0786 | 0.0755 | 0.0804 | 0.0745 | 0.0788 | 0.0774 | 0.0825 | 0.0756 | 0.0775 | 0.0763 |
| B | 0.0702 | 0.0786 | 0.0798 | 0.0794 | 0.0814 | 0.0813 | 0.0822 | 0.0818 | 0.0814 | 0.0775 | 0.0771 | 0.076 |
| C | 0.0709 | 0.0814 | 0.0784 | 0.086 | 0.0779 | 0.0786 | 0.0799 | 0.0776 | 0.0794 | 0.0761 | 0.075 | 0.076 |
| D | 0.0757 | 0.0799 | 0.0792 | 0.0802 | 0.0862 | 0.0799 | 0.0782 | 0.0791 | 0.0785 | 0.0839 | 0.08 | 0.0769 |
| E | 0.0716 | 0.0775 | 0.0754 | 0.0774 | 0.0847 | 0.0803 | 0.083 | 0.0758 | 0.0801 | 0.0791 | 0.0764 | 0.0802 |
| F | 0.0748 | 0.0772 | 0.0791 | 0.0804 | 0.0906 | 0.08 | 0.0808 | 0.0813 | 0.0784 | 0.0764 | 0.0808 | 0.0771 |
| G | 0.0738 | 0.0787 | 0.0787 | 0.08 | 0.082 | 0.0798 | 0.0815 | 0.0785 | 0.0816 | 0.0792 | 0.0761 | 0.071 |
| H | 0.073 | 0.082 | 0.0762 | 0.1198 | 0.0831 | 0.0814 | 0.0821 | 0.0787 | 0.0804 | 0.0753 | 0.0799 | 0.2075 |
第二轮初筛:共挑取了4块96孔板的微生物单克隆进行ELISA筛选(plate 05-08,每板另有2个对照克隆),详细实验数据见表格。
结论:第二轮初筛获得约90个潜在阳性单克隆
plate 05
| / | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | 0.2861 | 0.0619 | 0.1553 | 0.0571 | 0.0697 | 0.1173 | 0.2654 | 0.0631 | 0.1353 | 0.0567 | 0.0676 | 0.0561 |
| B | 0.174 | 0.0606 | 0.0587 | 0.0583 | 0.063 | 0.1602 | 0.056 | 0.0586 | 0.0623 | 0.1347 | 0.0615 | 0.056 |
| C | 0.0613 | 0.0912 | 0.0566 | 0.2031 | 0.0758 | 0.0636 | 0.0978 | 0.0585 | 0.1514 | 0.1922 | 0.0553 | 0.0561 |
| D | 0.063 | 0.1056 | 0.098 | 0.0726 | 0.0943 | 0.0655 | 0.061 | 0.0656 | 0.0557 | 0.1594 | 0.0552 | 0.0607 |
| E | 0.0605 | 0.1982 | 0.0564 | 0.1177 | 0.0627 | 0.0672 | 0.0604 | 0.0769 | 0.0608 | 0.0617 | 0.0634 | 0.063 |
| F | 0.0596 | 0.0609 | 0.0579 | 0.0616 | 0.0588 | 0.0642 | 0.0574 | 0.058 | 0.0568 | 0.1712 | 0.0987 | 0.0556 |
| G | 0.0565 | 0.0783 | 0.0568 | 0.2033 | 0.0826 | 0.0711 | 0.0556 | 0.1341 | 0.0553 | 0.1082 | 0.0548 | 0.0559 |
| H | 0.0827 | 0.2919 | 0.091 | 0.2727 | 0.0567 | 0.1121 | 0.0568 | 0.2702 | 0.0873 | 0.255 | 0.056 | 0.251 |
plate 06
| / | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | 0.0651 | 0.0566 | 0.0684 | 0.102 | 0.0653 | 0.0597 | 0.2466 | 0.0888 | 0.0683 | 0.2014 | 0.0899 | 0.0562 |
| B | 0.1529 | 0.0582 | 0.2096 | 0.057 | 0.0747 | 0.062 | 0.0555 | 0.0575 | 0.0657 | 0.0554 | 0.2129 | 0.055 |
| C | 0.0559 | 0.0596 | 0.2581 | 0.0582 | 0.0591 | 0.0569 | 0.1052 | 0.0566 | 0.0508 | 0.063 | 0.1112 | 0.0552 |
| D | 0.0972 | 0.2394 | 0.0759 | 0.0588 | 0.0582 | 0.0606 | 0.2575 | 0.0711 | 0.0555 | 0.0583 | 0.0557 | 0.0571 |
| E | 0.0579 | 0.0591 | 0.0578 | 0.0659 | 0.0974 | 0.0647 | 0.0575 | 0.2389 | 0.0557 | 0.0598 | 0.0582 | 0.0616 |
| F | 0.0591 | 0.0707 | 0.1009 | 0.0634 | 0.0777 | 0.0636 | 0.0592 | 0.0594 | 0.0572 | 0.0601 | 0.0648 | 0.0566 |
| G | 0.0574 | 0.1157 | 0.0565 | 0.0588 | 0.0554 | 0.1596 | 0.1966 | 0.0673 | 0.0539 | 0.1265 | 0.064 | 0.0552 |
| H | 0.0968 | 0.0674 | 0.0932 | 0.059 | 0.0571 | 0.0747 | 0.0819 | 0.0662 | 0.123 | 0.2302 | 0.1021 | 0.2622 |
plate 07
| / | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | 0.1742 | 0.0746 | 0.1105 | 0.0609 | 0.0627 | 0.0826 | 0.0851 | 0.0568 | 0.0548 | 0.1102 | 0.0547 | 0.0555 |
| B | 0.0589 | 0.1995 | 0.1255 | 0.1161 | 0.0573 | 0.1876 | 0.056 | 0.2677 | 0.055 | 0.0582 | 0.055 | 0.0552 |
| C | 0.0593 | 0.0579 | 0.0578 | 0.0703 | 0.0557 | 0.2025 | 0.0556 | 0.0669 | 0.0611 | 0.0789 | 0.0543 | 0.0573 |
| D | 0.0587 | 0.0983 | 0.1659 | 0.1289 | 0.0583 | 0.1227 | 0.0566 | 0.1582 | 0.0581 | 0.0674 | 0.0563 | 0.0657 |
| E | 0.0572 | 0.1654 | 0.0576 | 0.0716 | 0.2404 | 0.0701 | 0.0605 | 0.0668 | 0.2287 | 0.0694 | 0.0607 | 0.0641 |
| F | 0.059 | 0.0595 | 0.0618 | 0.0624 | 0.2407 | 0.0595 | 0.0716 | 0.106 | 0.0816 | 0.0667 | 0.0593 | 0.0594 |
| G | 0.0562 | 0.129 | 0.0572 | 0.1867 | 0.1882 | 0.0841 | 0.0554 | 0.071 | 0.0572 | 0.0628 | 0.0751 | 0.0562 |
| H | 0.0589 | 0.1726 | 0.0578 | 0.0689 | 0.0575 | 0.0637 | 0.0578 | 0.0673 | 0.0607 | 0.0664 | 0.0586 | 0.2658 |
plate 08
| / | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | 0.0583 | 0.0592 | 0.0575 | 0.0929 | 0.0585 | 0.1161 | 0.232 | 0.119 | 0.0644 | 0.0592 | 0.0568 | 0.1757 |
| B | 0.0556 | 0.124 | 0.0803 | 0.0577 | 0.0546 | 0.071 | 0.0571 | 0.0702 | 0.0573 | 0.0599 | 0.0591 | 0.1539 |
| C | 0.0794 | 0.0621 | 0.056 | 0.1091 | 0.0678 | 0.0594 | 0.2024 | 0.0608 | 0.0676 | 0.061 | 0.0579 | 0.0778 |
| D | 0.0661 | 0.067 | 0.0584 | 0.0731 | 0.1041 | 0.1036 | 0.0695 | 0.0728 | 0.0735 | 0.0703 | 0.1152 | 0.1019 |
| E | 0.0659 | 0.2459 | 0.0572 | 0.0663 | 0.0555 | 0.1708 | 0.0579 | 0.0677 | 0.0577 | 0.0778 | 0.0623 | 0.1592 |
| F | 0.0606 | 0.0586 | 0.0587 | 0.0571 | 0.0721 | 0.227 | 0.0957 | 0.0634 | 0.1765 | 0.0702 | 0.0636 | 0.0607 |
| G | 0.054 | 0.0666 | 0.0564 | 0.0617 | 0.0567 | 0.1463 | 0.0576 | 0.0697 | 0.0564 | 0.2615 | 0.1307 | 0.06 |
| H | 0.1496 | 0.1021 | 0.0728 | 0.0785 | 0.0577 | 0.0726 | 0.0561 | 0.1611 | 0.0783 | 0.1801 | 0.0595 | 0.2327 |
第一、二轮复筛:将初筛中获得的90余个单克隆挑选出来重新进行ELISA筛选(另有2个对照克隆),详细实验数据见下表格。
结论:复筛后保留32个潜在的阳性单克隆纳米抗体
| / | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A | 0.1263 | 0.0858 | 0.0863 | 0.0738 | 0.085 | 0.1102 | 0.0838 | 0.2779 | 0.2733 | 0.1157 | 0.0818 | 0.1336 |
| B | 0.1526 | 0.1888 | 0.0704 | 0.1205 | 0.0717 | 0.0801 | 0.0805 | 0.0833 | 0.0746 | 0.0753 | 0.2528 | 0.0771 |
| C | 0.0629 | 0.1208 | 0.0774 | 0.0876 | 0.2369 | 0.0768 | 0.1459 | 0.0728 | 0.114 | 0.0646 | 0.2313 | 0.0573 |
| D | 0.0796 | 0.113 | 0.0718 | 0.0755 | 0.1654 | 0.0958 | 0.0693 | 0.0693 | 0.0682 | 0.1419 | 0.0781 | 0.0584 |
| E | 0.0804 | 0.0692 | 0.0772 | 0.1529 | 0.068 | 0.0606 | 0.103 | 0.0683 | 0.0705 | 0.0798 | 0.1797 | 0.058 |
| F | 0.1057 | 0.1261 | 0.0785 | 0.0732 | 0.1378 | 0.0778 | 0.0995 | 0.1877 | 0.0657 | 0.3154 | 0.1249 | 0.0576 |
| G | 0.0908 | 0.079 | 0.0626 | 0.0795 | 0.1156 | 0.0841 | 0.0607 | 0.0888 | 0.0757 | 0.3253 | 0.1943 | 0.0612 |
| H | 0.0638 | 0.0943 | 0.094 | 0.0758 | 0.095 | 0.1342 | 0.2676 | 0.0871 | 0.093 | 0.0942 | 0.0728 | 0.2256 |
四、Sanger测序获得阳性VHH序列
- 将上述近32个单克隆进行Sanger测序,共得到32条有效的VHH基因序列。
- 经过翻译后,剔除错误的、重复的,最终得到23条序列不重复的VHH基因序列。
- 将23株单克隆纳米抗体进行重组表达纯化后,ELISA检测纳米抗体与IL18的亲和力,最终得到19株有效的纳米抗体